電気・情報生命工学科/電気・情報生命専攻
浜田 道昭准教授
Hamada Michiaki
| 略歴 | 2002年 東北大学大学院理学研究科数学専攻修士課程修了後、(株)富士総合研究所(現、みずほ情報総研(株))に入社。2009年 東京工業大学にて博士(理学)取得。2010年 東京大学大学院新領域創成科学研究科情報生命科学専攻 特任准教授を経て、2014年から現職。産業技術総合研究所客員研究員。日本バイオインフォマティクス学会(JSBi)理事。 |
|---|---|
| 主な担当科目 | バイオインフォマティクス、バイオインフォマティクス演習A-D、バイオインフォマティクス特論、Javaプログラミング |
| 主な著書 | 生命情報処理における機械学習 多重検定と推定量設計(機械学習プロフェッショナルシリーズ) (共著)、講談社、2015年 |
新しい研究分野、バイオインフォマティクス
バイオインフォマティクスは日本語では生命情報科学と呼ばれていますが、生物学・生命科学の問題を、主に情報科学の知識や技術を用いて解決しようとする研究分野です。計測機器・解析装置の性能が急速に向上するにつれて、例えばDNAやRNA、タンパク質などの配列情報が短時間で大量に得られるようになりました。2003年にヒトゲノムの全塩基配列=31億塩基対という膨大な情報を読み解く「ヒトゲノム計画」が完了したことをご存知の方も多いでしょう。この頃から、仮設を立て実験をして結果を得るという仮説駆動型で進められていた研究に加えて、蓄積された大量のデータを改めて分析し、新たな発見を目指すというデータ駆動型の研究も注目されるようになってきました。近年では10万人規模でのヒトゲノムデータ解読を目指すようなプロジェクトが動いています。また、ゲノムは1人につき1パターンですが、RNAやタンパク質は様々な組織や条件で多様な発現がありますから、これらを扱う研究プロジェクトで得られる情報量はさらに何倍にも膨れ上がります。このため、蓄積されたまま眠っているデータも多く、これを有効活用しようという動きが活発になってきているのです。ビッグデータを扱う研究開発分野は、宇宙物理や自然言語処理・画像解析などもありますが、「なぜ動物の体は複雑な作りをしているのか」「なぜ病気になるのか」「なぜ脳が超複雑な情報処理を行えるのか」といった、生命現象の「なぜ?」に対して、理学的な新しい発見を見込めるところが、バイオデータならではの面白さでしょうか。その発見が人類にとって有益になる可能性が大いにあり得る点にも、魅力があります。一方で、生物から得られるデータは、測定ノイズのみならず、実際の発現量のゆらぎなどで、ばらつきが出やすいです。いかにノイズを除去して本質を示すデータを取り出し、その奥にある生物学の特徴を見出すかという点には細心の注意が必要です。
出発は純粋数学
高校生のときに大学数学を学ぶ機会があり、小さな仮定=公理から全てが理論的に展開できる数学の世界に惹かれて、数学科に入学しました。数学分野では、学部生の間は特に、先人が導き出した数学理論をなぞるだけで精いっぱいです。修士課程でも、学生が新しい数学理論を導き出せる例は多くありません。そのような分野特有の状況にあって、ある限られた条件のもとではあるものの、「ある種の分布関数不等式を用いたティープリッツ及びハンケル作用素の解析」という新しい理論展開を、修士論文でまとめることができ、また、英文論文誌への掲載にも至りました。そのまま博士課程で数学を突き詰めていきたい気持ちもありましたが、修士課程で成果を出せたことで学問として数学と向かい合うことについては一区切りつけ、実社会で新しいことに挑戦してみようと就職を決めました。多様な経験ができそうなシンクタンク系を選び、入社後はまず、ナレッジマネジメントシステムの開発に携わることになりました。学生時代は「紙と鉛筆」の世界で生きていましたから、プログラミングの知識も皆無で、入社後に必死に勉強しました。給料をもらいながら新しい事を学ぶことができて、楽しかったですね。

写真1 浜田道昭先生
その後、「機能性RNAプロジェクト」という新エネルギー・産業技術総合開発機構(NEDO)の事業に、機能性RNAにかかわるバイオインフォマティクスの技術開発の担当者として参加することになり、そこで初めて分子生物学やバイオインフォマティクスといった分野を知ることになったのです。見ること聞くこと全てが初めてで、ここでも勉強三昧でしたが、物理や化学、情報、そして数学など、沢山の知識を駆使するバイオインフォマティクスは興味深く、のめり込んで行きました。同時に、学問を探求することも捨てきれず、社会人学生として博士課程に在籍し、博士号を取得しました。最初にお話ししたとおり、バイオインフォマティクスはまだ新しい専門分野ですから学術として確立させるためにできること、すべきことは多くあります。それを自分で成したいと思い、大学教員としての道を歩み始めました。
私自身は様々な分野の知識やスキルを楽しみながら身に付けてきましたので、学生にも楽しんで日々を過ごしてもらいたいですね。バイオインフォマティクスを題材として、普遍性のある、社会に出てからも役立つスキルを学んでもらえればと思っています。例えば、仮説を立て、筋道を立てて考え、結果を出すという方法論や、成果や考えを論理的に文章として書くことなどですね。研究室生活を通して、自立して物事を進められる力を養うことができれば、柔軟な対応ができますし、本当に興味を持てることに出会える可能性も高まります。

写真2 研究室では1人ひとりと向き合います
数学の知見を活かして、情報科学と生物学をつなぐ懸け橋に
バイオインフォマティクスには、理論的な厳密性よりも計算速度や実験結果との整合性を優先する「ヒューリスティック」なところがあります。私の場合は、数学的な素地を持っていますから、開発にあたっては数学的裏付けを取ること、結果の正しさを証明できる情報処理方法であることを意識して研究を進めています。例えば、RNA塩基対の2次構造(立体構造)を予測する方法はいくつか存在しますが、私たちの開発した「CentroidFold」は、現時点で最高精度を有し、塩基対の予測に関して理論的にも矛盾が少ない手法です(Hamada et al., “Prediction of RNA secondary structure using generalized centroid estimators”, Bioinformatics, 25, 465 (2009))。RNAは様々なタンパク質製造のためのレシピですが、後述のとおりタンパク質にならずにRNA自身が機能を持つnon-coding(ノンコーディング)RNAも数多く存在しています。そのようなnon-coding RNAの2次構造を正しく予測できれば、その機能の解明に近づきます。さらに、CentroidFoldを利用して、RNA自身を薬として働かせることができるRNAアプタマー配列の2次構造を予測する取組も進めています。また、RNAに限らず、データ駆動型の生命科学を実現するための基礎理論・技術の構築も行っています。様々な生命情報から生命現象の核心に迫る基盤情報技術として、バイオインフォマティクスが広く利用されるようになることを期待しながら、さらなる技術開発を進めています。
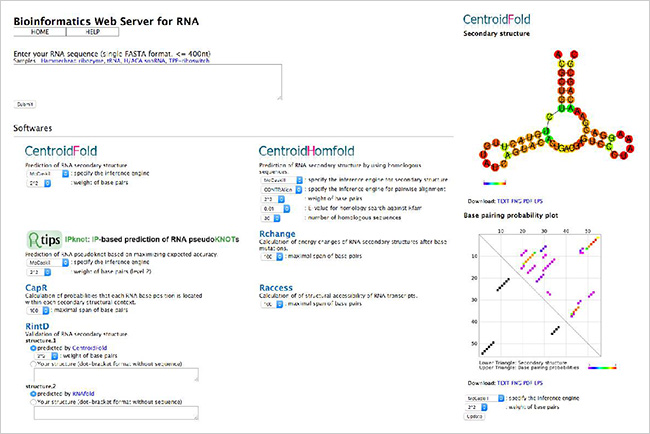
図1 CentroidFoldを含む数多くのRNA情報解析ツールを統合したWebサーバRtools
100年先まで利用されるアルゴリズムを
近年、タンパク質を作らず、RNAのまま働いているとされるlong non-coding RNA(lncRNA)が見つかっています。ヒトゲノムの中で、タンパク質をつくるRNAコードは2万種以上存在しています。一方、lncRNAはその3~5倍あると言われていますが、詳しい機能が分かっていません。その機能を情報科学的な見地から推定できる予測ツールを開発したいと考えています。RNAやタンパク質は単独で機能することは少なく、様々な相互作用の結果、実際の機能を発現していると考えられています。最近、どのRNAとRNAが相互作用しやすいか、あるいはしづらいのかといった予測を個々に特定していくツールの開発に成功しました。また、同様のことをRNAとタンパク質の相互作用に関しても行っています。さらには、すでに大量に蓄積されている全ゲノム、全RNA=トランスクリプトーム、全タンパク質=プロテオーム、後天的に変化・修飾されたゲノム=エピゲノム、生体分子間の相互作用=インタラクトームなどの階層化された性質の異なる生物データ(多層オミックスデータ)の各階層をターゲットにしたバイオインフォマティクス技術の開発も行っています。例えば、すでに蓄積されているゲノムワイドなエピゲノム情報から、その背後に存在する生物の構造を推定する方法の開発に成功しています(Hamada et al., “Learning chromatin states with factorized information criteria”, Bioinformatics 31, 2426 (2015))。さらに今後は、これら複数(あるいは全て)の階層を統合してデータマイニングを行うことを可能とする情報技術の開発を行いたいと思っています。
これらの研究開発を通して、将来は100年先まで利用される、本質的なバイオインフォマティクスの理論・アルゴリズム・ツールの開発に結び付けたいと思っています。バイオ分野のデータはそれぞれ性質が異なりかつノイズを多く含むため扱いが困難ですが、これらを一つのツールで統合して扱えるようになれば、これまでに見えていなかった情報、生命活動の本質が明らかになるのではないかと期待しています。開発したツールが、生命科学のブレイクスルーとなるような生物学的知見の発見につながれば、嬉しいです。同時に、研究室で学んだ学生たちが実社会のいろいろな分野で活躍するようになれば教員冥利につきますね。
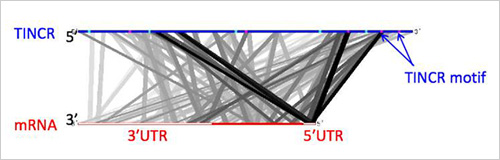
図2 相互作用の一例:皮膚の分化に必要となるlncRNA(TINCR)とmRNAの相互作用
聞き手・構成
武末出美(早稲田大学アカデミックソリューション)